El análisis profundo con IA revela antibióticos en el proteoma de las arqueas.
- 17 ago 2025
- 2 Min. de lectura
Actualizado: 19 ago 2025
GENÉTICA Y CIENCIA
Resumen Preparado por César Paz-y-Miño.
Articulo original:
Torres, M. D. T., Wan, F., & de la Fuente-Nunez, C. (2025). Deep learning reveals antibiotics in the archaeal proteome. Nature Microbiology. https://doi.org/10.1038/s41564-025-02061-0
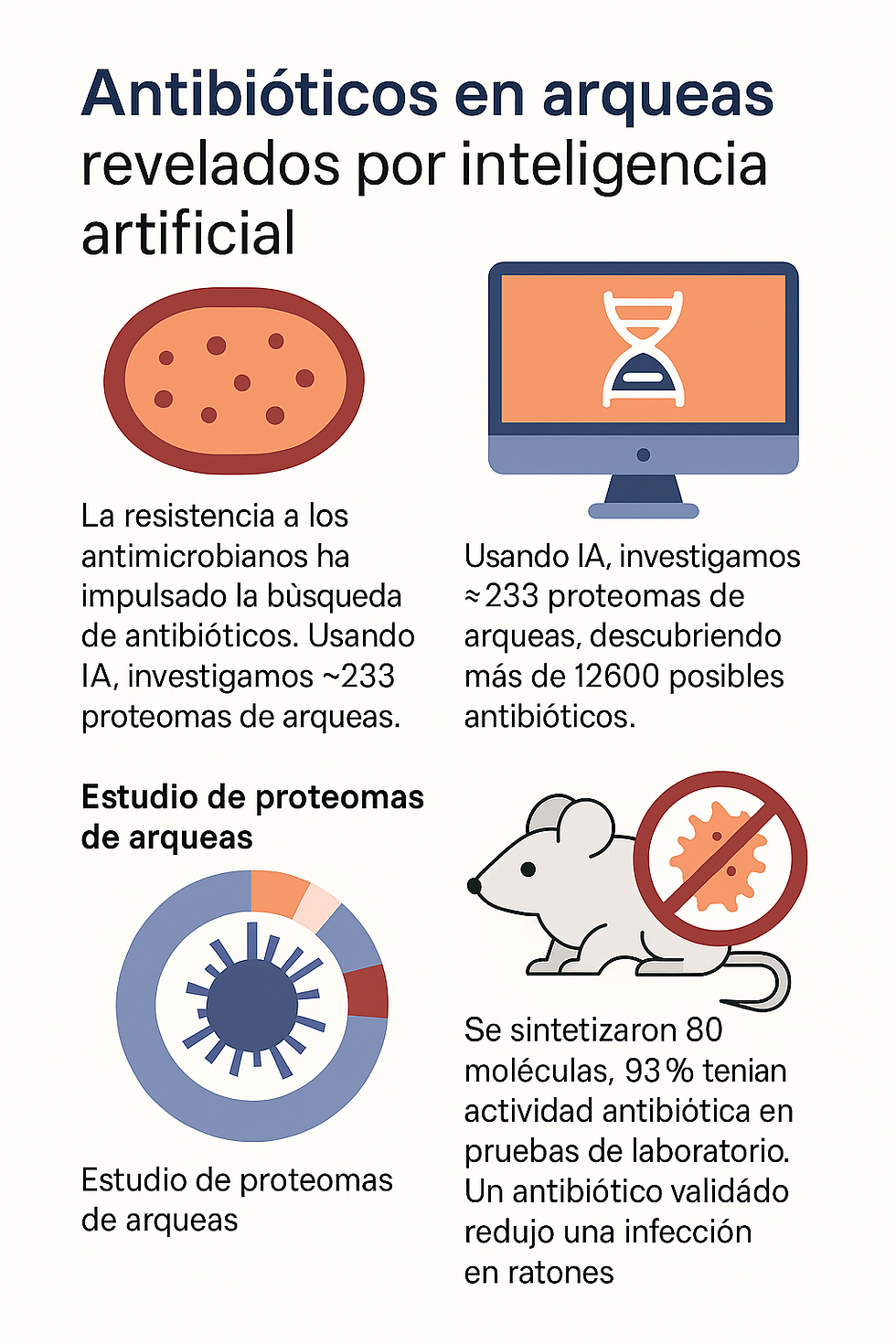
La resistencia antimicrobiana constituye una de las mayores amenazas para la salud global, ya que reduce drásticamente la eficacia de los antibióticos actuales. Tradicionalmente, la mayoría de antibióticos provienen de bacterias y hongos, pero las arqueas representan un reservorio casi inexplorado.
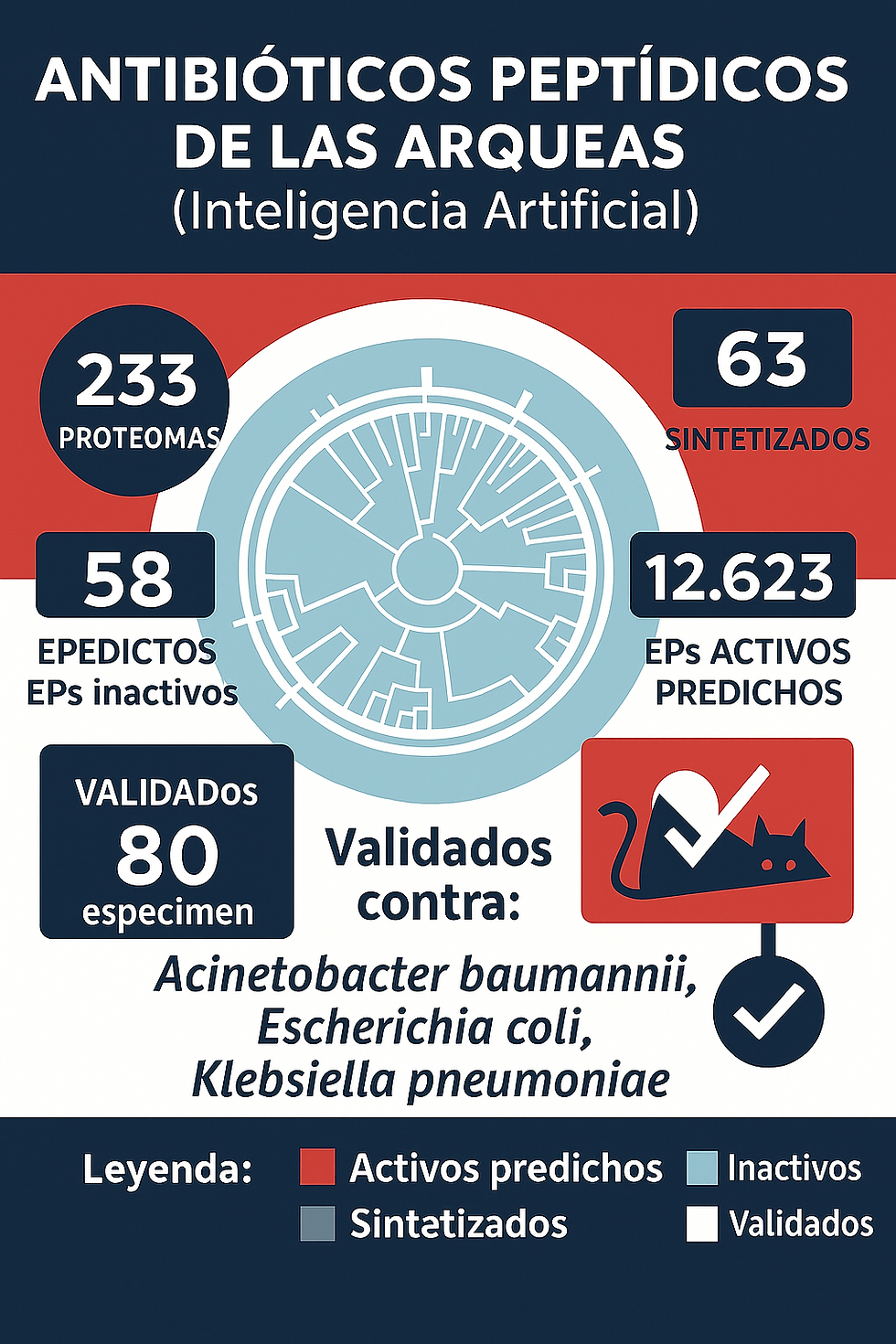
En este estudio, Torres, Wan y De la Fuente-Núñez aplicaron deep learning para analizar sistemáticamente el arqueoma (conjunto de proteomas de arqueas disponibles). A partir de 233 proteomas arqueales, el algoritmo predijo 12.623 péptidos con potencial actividad antimicrobiana, denominados arqueasinas (archaeasins).
Características principales de estos compuestos:
Poseen un perfil de aminoácidos único, distinto al de los péptidos antimicrobianos conocidos (AMPs), lo que sugiere mecanismos novedosos de acción.
Se sintetizaron experimentalmente 80 arqueasinas representativas.
El 93% mostró actividad antimicrobiana in vitro contra patógenos clínicos de alta preocupación: Acinetobacter baumannii, Escherichia coli, Klebsiella pneumoniae, Pseudomonas aeruginosa, Staphylococcus aureus y Enterococcus spp.
Entre los más prometedores, Arqueasina-73 logró reducir significativamente la carga bacteriana de A. baumannii en modelos murinos de infección, con una eficacia comparable a la del antibiótico de referencia polimixina B, actualmente usado como última línea contra bacterias multirresistentes.
Implicaciones científicas y médicas
Nueva fuente de antibióticos: las arqueas, organismos usualmente asociados a ambientes extremos, emergen como un reservorio inexplorado de moléculas antimicrobianas.
IA aplicada a la biomedicina: el uso de redes neuronales profundas permitió acelerar la identificación de candidatos, evitando búsquedas experimentales masivas y costosas.
Diversificación de estrategias terapéuticas: los péptidos arqueales poseen estructuras y composiciones únicas, lo que amplía el arsenal contra patógenos multirresistentes y reduce la posibilidad de resistencia cruzada.
Prueba de concepto validada in vivo: los resultados en modelos animales respaldan la aplicabilidad clínica futura, aunque aún se requieren estudios de toxicidad, estabilidad y farmacocinética.
Puerta a la bioprospección del arqueoma: con solo 233 proteomas analizados se obtuvieron más de 12.000 candidatos, lo que sugiere que el potencial es aún mayor si se exploran nuevas especies.
Enseñanza clave: este trabajo demuestra cómo la integración de genómica, proteómica, biología de arqueas y aprendizaje profundo puede revolucionar la búsqueda de antibióticos, aportando moléculas inéditas con eficacia comparable a fármacos establecidos y abriendo una vía prometedora para combatir la resistencia antimicrobiana.


Comentarios